DNA-technieken voor waterbeheerders
Dit Deltafact gaan in op de beschikbare DNA-technieken en de mogelijke toepassingen ervan in het water(kwaliteits)beheer.
|
Thema |
Waterkwaliteit, Diversen |
|
Tags |
|
|
Downloads |
1. INLEIDING
2. GERELATEERDE ONDERWERPEN EN DELTAFACTS
3. MOLECULAIRE TERMINOLOGIE
4. DNA REFERENTIEDATABANKEN
5. DE CYCLUS VAN EDNA IN DE OMGEVING
6. DE KETEN VAN (E)DNA BEMONSTERING
7. UITDAGINGEN OP GEBIED VAN DNA
8. ONTWIKKELINGEN OP HET GEBIED VAN DNA
9. CONCLUSIES EN AANBEVELINGEN
1. Inleiding
Steeds vaker komen termen als “DNA barcoding” en “environmental DNA” voorbij wanneer het gaat om de monitoring van waterkwaliteit. DNA technieken zijn de laatste jaren sterk doorontwikkeld, en bieden vandaag de dag verschillende mogelijkheden om te innoveren op het terrein van biomonitoring. Deze Deltafact gaat in op de diverse methoden die ten grondslag liggen aan de genetische biomonitoring. Er wordt stilgestaan bij de mogelijkheden die DNA biedt voor waterbeheerders, maar ook bij de aandachtspunten tijdens het hele proces van bemonstering tot interpretatie. De componenten die worden besproken in deze Deltafact zijn:
- Een beknopt overzicht van de beschikbare technieken en hun toepassingen;
- Het opzetten en gebruiken van referentiedatabanken;
- De herkomst en de afbraak van environmental DNA in de omgeving;
- De processen en te maken keuzen, vanaf bemonstering tot aan de resultaten;
- Uitdagingen en ontwikkelingen op gebied van DNA.
2. Gerelateerde onderwerpen en Deltafacts
Er bestaan geen Deltafacts direct gerelateerd aan de Deltafact ‘DNA technieken voor waterbeheerders’. De Deltafact ‘Remote sensing waterkwantiteits- en waterkwaliteitsbeheer’ gaat in op de toepassing van remote sensing bij de monitoring van waterkwaliteit, zoals die via spectrale beelden kan worden geïnterpreteerd.
3. Moleculaire terminologie>
Het gebruik van DNA technieken in de biologische monitoring bestaat uit twee componenten: identificatie en detectie. In beide gevallen zijn een aantal begrippen essentieel. DNA concentraties in een monster zijn vaak te laag om direct en betrouwbaar geanalyseerd te worden. Daarnaast is het DNA fragment dat wordt onderzocht maar een klein onderdeel van het totale genoom (de totale DNA informatie in een cel) van een organisme. Daarom wordt het DNA eerst vermeerderd (geamplificeerd) met PCR (Polymerase Chain Reaction, figuur 1), een enzymatisch proces waarin een klein fragment van het DNA wordt vermeerderd (ook wel amplificatie genoemd). Om een DNA fragment te kunnen amplificeren gebruikt men primers, speciaal voor dit doel gemaakte korte DNA fragmentjes die het amplificatieproces kunnen opstarten. De primers kunnen zo gekozen worden dat ze een kenmerkend stukje DNA van een organisme amplificeren (de DNA barcode). De term DNA barcoding werd in 2003 geopperd als methode om soorten gestandaardiseerd te kunnen identificeren (Hebert et al. 2003). De naam is ontstaan uit het gegeven dat een genetische code (een DNA sequentie) kan worden weergegeven in vier kleuren die de vier bouwstenen (de nucleotiden Adenine, Cytosine, Guanine en Thymine) van DNA representeren, hetgeen lijkt op een streepjescode (figuur 2).

Figuur 1. De PCR, een enzymatisch proces waarin DNA stapsgewijs wordt geamplificeerd: (1) DNA wordt enkelstrengs gemaakt in plaats van dubbelstrengs door het op te warmen, (2) de primer en het polymerase enzym binden aan het DNA, en het enzym start vanaf de primer en bouwt nucleotiden in om het DNA weer dubbelstrengs te maken (3). Daarna begint de cyclus weer vanaf het begin, waarbij steeds beiden helften van het DNA weer worden aangevuld naar dubbelstrengs DNA (4).
Na een PCR kan het vermeerderde DNA ontsleuteld worden in een proces dat sequencing wordt genoemd. Daarbij wordt de sequentie van het vermeerderde DNA afgelezen. De specifieke informatie in de DNA barcode wordt vervolgens gebruikt om soorten van elkaar te kunnen onderscheiden en op naam te brengen. In plaats van één enkele DNA barcode te bepalen van één enkel organisme, kan de techniek ook worden toegepast op een (meng)monster met daarin vele organismen (DNA metabarcoding). Voor DNA metabarcoding zijn er een aantal speciale sequencing technieken die geschaard worden onder de term Next-Generation Sequencing (NGS) of High-Throughput Sequencing (HTS). Tijdens dit proces is het mogelijk om in plaats van één enkele sequentie van een enkele soort, tot miljoenen sequenties van meerdere soorten tegelijkertijd af te lezen. Op die manier kan in één keer veel genetische informatie worden verkregen van een groot aantal organismen.

Figuur 2. DNA barcodes van verschillende Nederlandse zoetwatervissen
Als uitgangsmateriaal voor DNA metabarcoding kan men denken aan een biologisch monster dat volgens de traditionele meetmethoden of diatomeeënmonster. Een dergelijk monster kan als geheel verwerkt worden, waarbij organismen worden vermalen en al het aanwezige DNA uit de organismen wordt geanalyseerd (figuur 3, “bulk”). Minder destructieve methoden zijn ook mogelijk, door uitsluitend de ethanol te analyseren waarin de bulkmonsters veelal worden geconserveerd, en waarin DNA uit de organismen is vrijgekomen (figuur 3, “ethanol”). Bij het gebruik van DNA voor identificatie van organismen is een betrouwbare referentiedatabank nodig met DNA sequenties van goed geïdentificeerde soorten, waar de resultaten mee kunnen worden vergeleken (zie verder onder hoofdstuk 4). Op deze manier kunnen resultaten van bijvoorbeeld DNA metabarcoding worden gekoppeld aan soortnamen.
Naast de organismen zelf, kan ook gekeken worden naar DNA dat door organismen is losgelaten in de omgeving (zoals water of sediment), het zogenaamde environmental DNA (eDNA). Het gebruik van eDNA is de laatste tien jaar erg populair geworden (Ficetola et al. 2008). Met eDNA kan een beeld worden gevormd van soorten die zich in het water bevinden, zonder dat deze soorten fysiek verzameld hoeven te worden (figuur 3, “eDNA”). Micro-organismen, zoals diatomeeën en andere plankton zijn vaak in hun geheel aanwezig in een watermonster. Wanneer watermonsters worden verzameld voor de analyse van dit soort organismen, wordt echter vaak nog steeds over eDNA gesproken. Omdat het eDNA nagenoeg altijd uit een complexe mix van DNA van veel verschillende soorten bestaat, is het mogelijk om op een eDNA monster de metabarcoding techniek toe te passen. Op die manier kunnen soortgroepen of zelfs gemeenschappen die indicatief zijn voor de waterkwaliteit worden geanalyseerd, zoals vissen, dansmuggen, diatomeeën of bacteriën. Deze informatie kan deels gebruikt worden voor het invullen van KRW maatlatten, maar is ook inzetbaar voor bijvoorbeeld maatregel-effect analyses. In een ideale situatie worden alle gevonden DNA sequenties via de referentiedatabank naar soortnamen vertaald. De praktijk wijst uit dat er nog veel organismen in het water zijn waarvan geen referentie sequenties beschikbaar zijn. Het komt dan ook regelmatig voor dat DNA sequenties in een analyse geen identificatie tot op soortniveau krijgen (maar mogelijk wel op een hoger taxonomisch niveau, zie “Bioinformatica” in hoofdstuk 6). Omdat DNA barcoding methoden echter ontwikkeld zijn op basis van het feit dat de DNA barcodes in de meeste gevallen uniek zijn voor één soort, kan dergelijke informatie nog steeds van nut zijn om verschillen tussen locaties of tussen metingen in de tijd te onderzoeken (zie bijvoorbeeld “Taxonomie-vrije methoden” in hoofdstuk 8).

Figuur 3. Overzicht van de verschillende methodieken om met behulp van DNA inzicht te krijgen in de gemeenschap van organismen in een waterlichaam, zoals bijvoorbeeld macrofauna, en welk type monster geschikt is voor iedere techniek
Een PCR kan bij de eDNA analyse ook worden ingezet voor de amplificatie van DNA van één specifieke doelsoort. Door de primers dusdanig specifiek te maken dat ze enkel passen op het DNA van een enkele soort, is het mogelijk om uit een mengmonster van meerdere soorten (zoals een eDNA monster), alleen het DNA te amplificeren van deze doelsoort. Deze speciale primers kunnen worden ingezet bij een kwantitatieve PCR (quantitative PCR of qPCR). Hiermee kan een specifieke soort gedetecteerd worden in een waterlichaam, en kan ook een uitspraak worden gedaan over verschillen in de hoeveelheden aangetroffen DNA van de betreffende doelsoort tussen verschillende locaties. De soort-specifieke qPCR is een zeer geschikte methode om eDNA monsters gericht te testen op de aanwezigheid van beschermde soorten, invasieve exoten of ziekteverwekkers. Een variant op de qPCR is de digital droplet PCR (ddPCR). Deze methode is nauwkeuriger dan de reguliere qPCR en bovendien in staat om lagere concentraties van DNA van de doelsoort te detecteren (Doi et al. 2015).
4. DNA referentiedatabanken
De toepassing van DNA barcoding technieken baseren zich op het feit dat de DNA barcode uniek is voor een soort. Met andere woorden, de sequentie verschilt tussen soorten, maar is gelijk tussen individuen van één soort. Wanneer twee soorten geen of slechts weinig variatie vertonen in de sequentie van het gekozen DNA fragment, zijn ze niet of moeilijk van elkaar te onderscheiden. Aan de andere kant, als sequenties te veel variatie vertonen tussen individuen binnen een soort, is het lastig om een referentie sequentie te bepalen die representatief (altijd hetzelfde) is voor alle individuen van die soort. Om optimaal gebruik te maken van DNA gegevens is het belangrijk om de DNA profielen te kunnen koppelen aan taxonomische (soort)namen. Via deze soortnamen weten we namelijk welke soorten er op een bepaalde plek zitten en kunnen ecologische interpretaties gedaan worden die moeten leiden tot meer inzicht in het bemonsterde systeem. Om de aangetroffen DNA sequenties te kunnen herleiden tot taxonomische (soort)namen is een betrouwbare DNA referentiedatabank nodig. Hierin liggen de sequenties opgeslagen van goed geïdentificeerde exemplaren van soorten, die bij voorkeur als controleerbare voucher specimen in een toegankelijk collectie zijn opgenomen. Op deze wijze kan bij twijfel over identificaties worden teruggegrepen op die exemplaren, of kunnen gewijzigde taxonomische inzichten worden doorgevoerd. Dit houdt de referentiedatabank actueel.
Wereldwijd wordt gewerkt aan een zo betrouwbaar mogelijke DNA referentiebibliotheek, een initiatief dat is ontstaan in het Canadese Guelph, waar Paul Hebert in 2003 het voortouw nam om van zoveel mogelijk plant- en diersoorten ter wereld DNA barcodes te bepalen (Hebert et al. 2003). Al deze barcodes worden samengebracht in een internationale databank, de “Barcode of Life Database” (BOLD; www.boldsystems.org). De afgelopen jaren zijn grote voucher collecties die ten grondslag lagen aan de DNA barcodes samengebracht bij diverse natuurhistorische musea in de wereld, waaronder het Naturalis Biodiversity Center in Leiden.
Een goed gevalideerde referentie is van essentieel belang om gefundeerde uitspraken te kunnen doen over de aanwezigheid van soorten op basis van DNA. Het is daarbij belangrijk om een referentiedatabank zo veel mogelijk te laten aansluiten op de lokale biodiversiteit. Gebruik van publieke data kan daarbij een risico vormen omdat DNA referentiesequenties uit andere landen of werelddelen zeker niet altijd representatief zijn voor de lokale populaties van soorten. Daarnaast houden verschillende landen soms verschillende taxonomische indelingen of naamgevingen aan, waardoor niet altijd een correcte koppeling gemaakt kan worden met bijvoorbeeld de KRW maatlatten. Naast BOLD zijn er nog diverse andere publieke referentiebibliotheken online beschikbaar. BOLD is beperkt tot dieren en planten, en maakt gebruik van een beperkte set aan DNA barcodes. Meer informatie is te vinden op GenBank (www.ncbi.nlm.nih.gov/genbank), maar de betrouwbaarheid van veel informatie hierin is twijfelachtig, omdat kwaliteitscontrole voor deze referentiedatabank ontbreekt. Voor bepaalde groepen organismen zijn er specialistische referentiedatabanken te vinden, bijvoorbeeld de Silva databank voor met name bacteriën (https://www.arb-silva.de/). Buiten de publieke databanken hebben veel instellingen en bedrijven ook hun eigen referentiedatabanken, die niet altijd publiek toegankelijk zijn.
5. De cyclus van eDNA in de omgeving
Voor een optimale bemonstering van eDNA in een waterlichaam is het belangrijk om te weten wat de oorsprong en het uiteindelijke lot van eDNA in de omgeving is (figuur 4) (Barnes & Turner 2015). Deze kennis is nodig bij een gedegen ecologische interpretatie van het al of niet aantonen van eDNA sporen.
Figuur 4 (rechts) De belangrijkste processen in de cyclus van eDNA in de omgeving: (1) oorsprong, (2) staat, (3) transport en (4) afbraak
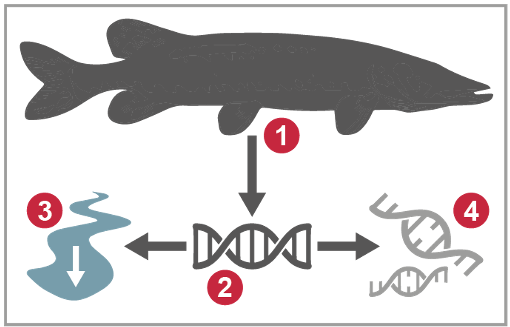
Hoe detecteerbaar is een soort via eDNA, en in hoeverre duidt de detectie van eDNA van een soort ook op de daadwerkelijke recente en lokale aanwezigheid van die soort? Organismen laten op verschillende manieren biologisch materiaal, met daarin DNA, achter in de omgeving. Een groot deel daarvan is afkomstig van dode huidcellen en ander materiaal dat wordt afgeschud. De voornaamste bron van eDNA wisselt per soortgroep.
Oorsprong van DNA. Uit de relatief succesvolle experimenten met eDNA detectie van zowel vissen als amfibieën kan geconcludeerd worden dat de slijmlaag die deze dieren uitscheiden als verdedigingsmechanisme tegen ziekteverwekkers een belangrijke bron is van eDNA (Ficetola et al. 2008, Jerde et al. 2011). Organismen die veel water filteren, waaronder mosselen en andere tweekleppigen, laten organisch materiaal vrij via het uitgescheiden filtraat, maar introduceren via het kuitschieten ook veel DNA in de omgeving (Sansom & Sassoubre 2017). Voor veel insecten geldt dat het afschudden van celmateriaal beperkter is dan bij vertebraten, en deels gelimiteerd tot de larvale stadia die vervellen en uiteindelijk verpoppen. Zo zijn dansmuggen relatief goed te detecteren middels het DNA dat vrijkomt uit larven- en poppenhuidjes (Bista et al. 2017). Daarnaast vormen uitwerpselen van veel dieren ook een uitstekende bron van eDNA, zowel hun eigen DNA als het DNA van de organismen in hun dieet.
Staat van DNA. Hoewel de associatie met vrij-zwevend DNA in de waterkolom vaak wordt gemaakt wanneer over eDNA wordt gesproken, is uit onderzoek gebleken dat het meeste “vrije” DNA toch nog gebonden zit in cellen en cel-resten (Turner et al. 2014, Wilcox et al. 2015). Echt vrij-zwevend DNA is weliswaar een onderdeel van het totale eDNA, maar is minder stabiel omdat het direct blootgesteld wordt aan de biologische en chemische processen die DNA afbreken in de waterkolom.
Transport van DNA. De staat van het eDNA, gebonden in cellen en cel-resten, heeft ook invloed op het transport van eDNA door een waterlichaam. Er is nog weinig onderzoek gedaan naar de diffusie en/of dispersie afstand van eDNA in stilstaande wateren, maar door het feit dat DNA zich nog in cellen bevind of aan deeltjes bindt, zal het de neiging hebben om neer te slaan op de bodem. In stromende wateren is aangetoond dat eDNA soms tot op enkele kilometers stroomafwaarts nog detecteerbaar was (Civade et al. 2016, Deiner et al. 2016,). Bij de bemonstering van eDNA is het essentieel om rekening te houden met beweging van eDNA binnen een waterlichaam.
Afbraak van DNA. eDNA is maar gedurende korte tijd aanwezig in de waterkolom. Onderzoek aan de persistentie van eDNA in water voor een aantal verschillende soorten amfibieën en vissen heeft uitgewezen dat eDNA enkele dagen tot soms maximaal twee weken aanwezig is in het water (Dejean et al. 2011, Thomsen et al. 2012). Fysisch-chemische en biologische factoren beïnvloeden de afbraaksnelheid van DNA. Zo blijft eDNA minder lang detecteerbaar in warm water, bij veel zonlicht, of bij hoge bacteriële activiteit (Strickler et al. 2015). Dit betekent dat de aanwezigheid van eDNA duidt op een recente aanwezigheid van de soort. In het sediment kan het eDNA veel langer bewaard blijven, als de condities daar geschikt voor zijn (Turner et al. 2015), en sedimentmonsters zijn daarom minder geschikt voor het aantonen van recente aanwezigheid van soorten.
6. De keten van (e)DNA bemonstering
Locatie selectie en monstername. Een bemonsteringsstrategie voor eDNA moet vanzelfsprekend goed worden afgestemd op de ecologische vraagstelling. Voor de traditionele monitoring bestaan diverse protocollen, zoals de werkvoorschriften die zijn vastgelegd in het Handboek Hydrobiologie (STOWA 2014). Voor bijvoorbeeld macrofauna monsters die traditioneel verzameld worden, maar daarna via DNA technieken worden geïdentificeerd, kunnen de gebruikelijke werkvoorschriften worden gevolgd. Voor het selecteren van locaties voor eDNA bemonstering kan men die werkvoorschriften eveneens volgen, waarbij het vooral belangrijk is om een waterlichaam voldoende ruimtelijk te bemonsteren. DNA sporen kunnen, zeker in stilstaande wateren, een vrij lokaal signaal opleveren, en soorten die zich weinig verspreiden door een waterlichaam kunnen daardoor makkelijk gemist worden als de eDNA bemonstering slechts op één punt gedaan wordt. Vaak wordt gebruik gemaakt van zogenaamde mengmonsters, waarbij water van verschillende locaties binnen een waterlichaam wordt gecombineerd tot een monster van een groter volume. Er is nog veel onderzoek gaande naar optimalisatie van de DNA bemonsteringsstrategie, en tot op heden is er geen “uniforme” of “beste” methode.
Uiteraard zullen de kosten ook meewegen in de keuze voor een bemonsteringsstrategie. In sommige gevallen zullen mengmonsters afdoende zijn, in andere gevallen is het beter om monsters van verschillende locaties separaat te houden, wanneer men naar verspreidingspatronen wil kunnen kijken. Ook de diepte van bemonstering kan variëren, van bovenin de waterkolom tot vlak bij de bodem, afhankelijk van de doelsoorten en het watertype. Naast de locatie en ruimtelijke bemonstering daarbinnen, is het ook belangrijk om op het juiste moment in het jaar een eDNA monster te nemen, bijvoorbeeld waar het ongewervelden betreft. De eDNA bemonstering van bepaalde insecten, zoals libellen of dansmuggen, zal minder efficiënt zijn als de larven niet meer in de waterkolom aanwezig zijn. Ook voor micro-organismen zijn er redelijk grote verschillen waar te nemen tussen monsters die slechts enkele weken uit elkaar zijn genomen. Dit maakt eveneens duidelijk dat het moment waarop een monster genomen wordt goed meegewogen moet worden in de bemonsteringsstrategie (Beentjes et al. 2019a).
Ongeacht de keuzes die gemaakt worden omtrent de bemonsteringsstrategie en technieken, is het van belang dat aandacht wordt besteed aan het voorkomen van kruisbesmettingen tussen monsters. Zeker waar het eDNA betreft, is het essentieel om materialen die worden hergebruikt tussendoor te reinigen, zodat er geen DNA vanuit een vorige locatie wordt meegenomen naar de volgende locatie. Hiermee wordt voorkomen dat er zogenaamde “vals positieve” signalen worden aangetroffen, waarbij soorten worden gedetecteerd in het DNA monster die eigenlijk niet aanwezig waren op de betreffende monster locatie. Een manier om verontreiniging in het veld uit te sluiten is door blanco monsters te nemen tijdens het veldwerk. Dit zijn monsters genomen uit gezuiverd water, waarin zeker geen DNA voorkomt.
Bewaren van monsters. Afhankelijk van het type monster zijn er diverse methoden om deze te bewaren. Verzamelde macrofauna of plankton bulk kan het beste worden opgeslagen op een hoog percentage ethanol (70-96%). Deze ethanol kan ook gebruikt worden als bron van DNA (figuur 3), omdat organismen in de ethanol veel DNA vrijlaten. Het eDNA uit watermonsters wordt idealiter in het veld gefilterd, waarna het filter in een conserveringsmedium bewaard kan worden tot het moment van de extractie. Als alternatief kan water worden gecombineerd met een conserveringsmedium om filtratie in het veld te vermijden (Williams et al. 2016, Yamanaka et al. 2017). Bewaren van niet geconserveerde en/of niet gefiltreerde watermonsters voor de extractie van eDNA is altijd suboptimaal, omdat afbraakprocessen ook na bemonstering doorgaan. Invriezen van water heeft ook een nadelig effect op de DNA opbrengst.
DNA extractie. Bij de DNA extractie wordt het DNA vrijgemaakt uit het verzamelde organisch en omgevingsmateriaal. Filters waarin eDNA is opgevangen of watermonsters waarin DNA is geprecipiteerd bevatten naast DNA nog restanten van cellen (zie “Staat van DNA” in hoofdstuk 5). Hetzelfde geldt uiteraard ook voor bulk monsters die worden vermalen. Tijdens de DNA extractie worden intacte cellen enzymatisch opengebroken, zodat alle cel-gebonden DNA in oplossing komt. Doordat DNA negatief geladen delen bevat, kan het DNA middels een chemisch proces worden gescheiden van de andere componenten, meestal door het tijdelijk te binden aan een speciale matrix. Aan het einde van het extractie proces resteert een DNA extract met zuiver DNA. Er zijn diverse soorten DNA extractiemethoden beschikbaar, onder andere in kant-en-klare, commercieel verkrijgbare kits. Verschillende methoden hebben elk hun voor- en nadelen, zowel voor maximale DNA opbrengst en extractie-efficiëntie, als voor gebruiksgemak, opschaalbaarheid en kosten per monster.
Amplificatie. Normaliter zijn DNA concentraties die verkregen worden uit een extractie vrij laag, en de gebruikelijke technieken voor sequentie analyse zijn niet gevoelig genoeg om met deze zeer lage DNA concentraties betrouwbare resultaten te verkrijgen in een sequentie analyse. Het vrijgekomen DNA uit de extractie wordt daarom vrijwel altijd geamplificeerd voor verdere verwerking. Meestal wordt alleen de gekozen DNA barcode regio geamplificeerd. Een amplificatie reactie (de PCR) is een exponentieel proces. Dat wil zeggen dat de gekozen barcode in elke cyclus van de reactie wordt verdubbeld (figuur 1). Meestal bevat een PCR ongeveer 20 tot 30 cycli, en in een ideale situatie zou een DNA fragment dus moeten leiden tot ongeveer een miljoen (220) c.q. een miljard (230) kopieën.
Vanaf de start van de DNA extractie vinden alle werkzaamheden idealiter plaats in speciaal ingerichte laboratoria. Wanneer veel wordt gewerkt met DNA in een laboratorium, moeten maatregelen getroffen worden om te voorkomen dat er kruisbesmettingen plaatsvinden tussen verschillende monsters (om vals positieven te voorkomen). Essentieel is een scheiding tussen zogenaamde pre-PCR (voor de PCR) en post-PCR (na de PCR) werkzaamheden. PCR producten zijn vaak relatief klein qua omvang (een DNA barcode is maar een klein stukje DNA) en in zeer hoge concentraties aanwezig, en kunnen daardoor via aërosolen makkelijk de originele DNA extracten, die meestal lage concentraties DNA bevatten, besmetten. Om kruisbesmetting tussen PCR producten zo veel mogelijk te voorkomen kunnen maatregelen worden getroffen, zoals het gebruik van filterpuntjes voor het pipeteren, zodat er geen aërosolen in de pipetten terecht kunnen komen die worden overgedragen naar andere monsters. Het is zeer moeilijk om alle bronnen van mogelijke kruisbesmetting van te voren volledig uit te sluiten, en het is dus essentieel om in het laboratoriumproces de nodige controle monsters mee te laten lopen waarmee mogelijke kruisbesmettingen inzichtelijk gemaakt kunnen worden zodat daarmee eventueel kan worden gecorrigeerd. Voor de PCR is er een controle in de vorm van een amplificatie-blanco, waarin geen DNA aanwezig is. DNA dat in deze monsters gevonden wordt is dus afkomstig van kruisbesmettingen tussen monsters in het lab. Ook kunnen positieve controles worden meegenomen om te controleren of de verschillende stappen in het laboratorium wel efficiënt zijn verlopen. Zo kan inzichtelijk gemaakt worden of een negatief resultaat (geen PCR product) veroorzaakt wordt door de afwezigheid van DNA (van een doelsoort), of dat de amplificatie reactie niet heeft gewerkt. Het is dan wel aan te raden om een positieve controle te kiezen die geen soorten bevat die ook verwacht worden in de te analyseren monsters, in verband met mogelijke contaminatie vanuit de positieve controle naar de daadwerkelijke monsters.
Kwantitatieve PCR. Een speciale manier van PCR is de kwantitatieve PCR (de qPCR en ddPCR zoals beschreven in hoofdstuk 3). Hierin worden primers gebruikt die specifiek het DNA van één bepaalde soort (of een soortgroep) amplificeren. De meest basale methode van kwantitatieve PCR werkt met toevoeging van een kleurstof die een fluorescent signaal afgeeft wanneer deze is gebonden met dubbelstrengs DNA. Aangezien er in iedere PCR cyclus een verdubbeling plaatsvindt van het DNA (figuur 1), zal er steeds meer dubbelstrengs DNA aanwezig zijn, en dus steeds meer fluorescent signaal. Het signaal wordt na iedere cyclus gemeten, en uit de intensiteit van het signaal kan wordt berekend wat de oorspronkelijke concentratie van DNA van de doelsoort was. Een kwantitatieve PCR kan nog specifieker en gevoeliger gemaakt worden door het gebruik van een fluorescente “probe”, waarbij alleen een fluorescent signaal wordt afgegeven bij binding aan het geamplificeerde DNA (op basis van de specifieke sequentie van de doelsoort).
Next-Generation Sequencing. Na de amplificatie van target soorten of soortgroepen via de PCR, moeten de sequenties worden afgelezen om de basenvolgorde te bepalen. Omdat het vaak mengsels betreft van veel verschillende soorten, en dus verschillende sequenties, moeten de geamplificeerde DNA fragmenten individueel uitgelezen worden. Next-generation sequencing machines zijn ontworpen om miljoenen DNA fragmenten tegelijkertijd uit te lezen. Vaak worden meerdere monsters gecombineerd op een enkele “run” in een NGS apparaat. Door de primers in de PCR in de voorbereiding van NGS te voorzien van unieke labels en elk monster met een unieke combinatie van gelabelde primers te amplificeren, is het mogelijk de monsters van meerdere locaties samen te voegen, en ze later op basis van de labels weer van elkaar te kunnen scheiden.
Bioinformatica. De keuzes die gemaakt worden bij de verwerking van NGS data zijn net zo belangrijk als de keuzes die gemaakt worden voor het bemonsteren in het veld, of de verwerking in het laboratorium. De ruwe gegevens die worden gegenereerd door het NGS apparaat zijn namelijk onbruikbaar voor analyses. Allereerst moeten er sequentiefouten en onnauwkeurigheden verwijderd worden. Aan elke toekenning van een base (A, C, G of T) tijdens het sequencen is door de machine een kwaliteitsscore meegegeven, welke aangeeft wat de zekerheid is dat die base juist is. Op basis van deze kwaliteitsscore kunnen sequenties van lage kwaliteit worden verwijderd uit de ruwe data.
Op het moment dat de ruwe data zo veel mogelijk opgeschoond zijn, kunnen de overgebleven sequenties worden vergeleken met de referentiedatabank. Echter, bij een opbrengst van miljoenen sequenties kan het proces van vergelijken met de referentie barcodes veel tijd in beslag nemen. Omdat in de meeste gevallen niet miljoenen soorten aanwezig zijn, is het daarom logisch om eerst alle DNA sequenties die op elkaar lijken bij elkaar te clusteren (figuur 5). Het clusteren van sequenties gaat over het algemeen op basis van de overeenkomst van de basenvolgorde, de clusters die overblijven worden vaak MOTU’s (molecular operational taxonomic units, ook wel OTU’s) genoemd. Omdat de oorspronkelijke sequenties nog altijd informatie bevatten over het monster waaruit zij afkomstig zijn (doordat deze waren voorzien van unieke labels), kan na het clusteren een zogenaamde MOTU-tabel worden gemaakt, een kruistabel van MOTU’s en monsters, waarin is te zien hoeveel sequenties van elke MOTU aanwezig waren in elk monster.
Voor het vergelijken met de referentiedatabank wordt meestal een BLAST (basic local alignment search tool) algoritme gebruikt, die de MOTU’s met alle sequenties uit de referentiedatabank vergelijkt, en een match zoekt met de sequentie die het meeste overeenkomt met de MOTU. In de meeste analyses worden aan deze matches wel bepaalde eisen gesteld, zo is het gebruikelijk een ondergrens van bijvoorbeeld 98% overeenkomst in te stellen, zodat er geen positieve matches plaatsvinden als sequenties (in dit geval) meer dan 2% afwijken van de referentie.

Figuur 5. Bioinformatica stappen voor de verwerking van ruwe NGS data. In dit voorbeeld zijn de unieke labels die via de PCR zijn ingebouwd aangegeven met letters A, B en C. Na het clusteren kan worden gesplitst op deze labels, om samples A, B en C individueel te analyseren.
In de meeste situaties is er namelijk ongeveer minstens 2% verschil tussen verwante soorten. Door een grens van 98% aan te houden, wordt voorkomen dat een match wordt gevonden met een andere, verwante soort (een vals positief signaal). Het komt regelmatig voor dat referentiedatabanken geen volledige dekking hebben van alle mogelijke soorten, en er dus MOTU’s overblijven zonder identificatie. Het is voor deze MOTU’s echter nog steeds mogelijk om er in plaats van een identificatie op soortniveau een identificatie van een hoger taxonomisch niveau (bijvoorbeeld geslacht of familie) aan toe te kennen, middels een “lowest common ancestor” (LCA) analyse (Beentjes et al. 2019b).
Analyse. Afhankelijk van de vraagstelling bij het onderzoek, kan een soortenlijst verkregen uit de DNA metabarcoding al voldoende zijn, of kunnen er, net als met data verkregen uit andere methodieken, diverse biodiversiteits-analyses uitgevoerd worden. Het is belangrijk om er bij het werken met op DNA gebaseerde gegevens rekening mee te houden dat de interpretatie van resultaten wel enige kennis vereist van de methoden die er achter liggen. Voor de kwantitatieve soort-specifieke resultaten verkregen met qPCR en ddPCR geldt dat een positief dan wel negatief signaal niet direct is te vertalen naar “aanwezigheid/afwezigheid”, maar dat eigenlijk gesproken moet worden van “aangetoond/niet aangetoond”. Het kan immers (net als bij morfologische monitoring) nog steeds mogelijk zijn dat een soort wel degelijk aanwezig is, maar dat het eDNA in lage hoeveelheden in het water zit en de trefkans heel laag is. Ook kan een DNA concentratie niet direct worden vertaald naar een abundantie in termen van biomassa of aantallen individuen. Voor metabarcoding geldt hetzelfde, de soort kan in te lage dichtheden in het water zitten of te weinig DNA achterlaten om met deze analyse opgepikt te kunnen worden. Daarnaast kan de “afwezigheid” van een soort tijdens een metabarcoding analyse ook worden veroorzaakt doordat de gebruikte primers niet of minder geschikt zijn om net die soort te kunnen amplificeren (“PCR bias”). Primers voor metabarcoding zijn namelijk niet soort-specifiek maar ontworpen om meerdere soorten of soortgroepen tegelijkertijd te amplificeren.
7. Uitdagingen op gebied van DNA
Vals negatieven en positieven. Naast de eerder genoemde discussies rond “aanwezigheid/afwezigheid” (feitelijk “aangetoond/niet aangetoond”, zoals benoemd onder “Analyse” in hoofdstuk 6), en het gebruik van abundantie-data voor NGS analyses (zie “Abundantie” later in dit hoofdstuk) zijn er nog een aantal zaken om rekening mee te houden bij het werken met eDNA. De “vals negatieve” signalen die gevonden kunnen worden zijn zoals eerder besproken mogelijk situaties waarin eDNA in te lage concentraties aanwezig is voor de gebruikte methoden, of waar de gebruikte primers niet geschikt zijn. Voor metabarcoding kan ook de PCR bias er toe leiden dat bepaalde soorten verloren raken in de achtergrond omdat de primers een betere binding hebben met het DNA van andere soorten. Zeldzame soorten kunnen daarnaast bij NGS ook simpelweg niet gedetecteerd worden als er veel DNA van andere soorten aanwezig is, omdat er ook zonder PCR bias nog een competitie is tussen de DNA moleculen voor de PCR chemicaliën, en er (afhankelijk van de geanalyseerde hoeveelheid DNA materiaal) bij de sequencing een maximaal aantal DNA sequenties afgelezen wordt. Deze zogenaamde “sequencing diepte” wordt deels bepaald door beperkingen van gekozen NGS machines, maar is ook een kostentechnische overweging (meer sequenties per monster analyseren is namelijk duurder).
Daarnaast zijn er ook zogenaamde “vals positieven”, zoals al eerder benoemd. Deze worden voornamelijk veroorzaakt door contaminatie tussen monsters, hetgeen zowel in het veld bij de bemonstering als in het lab bij de verwerking kan plaatsvinden. Ook kan DNA worden aangetroffen dat op een andere manier ter plaatse is gekomen dan direct van het betreffende organisme, denk bijvoorbeeld aan uitwerpselen van watervogels.
Referentiedatabanken. Naast de bron van het eDNA kan ook de gebruikte referentiedatabank leiden tot vals negatieve en positieve signalen. Het is belangrijk dat een referentiedatabank wordt samengesteld uit betrouwbare sequenties, een foute identificatie kan er namelijk toe leiden dat een foutieve soortnaam wordt toegevoegd aan de gevonden biodiversiteit. Gaten in de databank leiden daarentegen tot vals negatieven, waar een soort niet wordt gevonden omdat er geen referentie is om de eDNA sequentie tegen te matchen. Ook voor goed-bestudeerde groepen zoals de Europese zoetwater-macrofauna zijn er nog steeds lacunes in de publieke databases, die er in sommige gevallen toe leiden dat DNA technieken niet optimaal benut kunnen worden (Weigand et al. 2019). Zeker NGS analyses waarbij het doel is om een soortenlijst op te leveren staan of vallen met de kwaliteit en compleetheid van de gebruikte DNA referentiedatabank. Zoals aangestipt in hoofdstuk 4 zijn niet alle publieke databanken even betrouwbaar, en wordt in een ideale situatie gebruik gemaakt van referentie DNA afkomstig uit gevouchered materiaal.
Genetische variatie. Ondanks de aanname van de eerder genoemde 2% variatie tussen verwante soorten (zie “Bioinformatica” in hoofdstuk 6) zijn er wel degelijk soorten waarbinnen veel genetische variatie bestaat tussen verschillende populaties. Dit geldt zeker voor populaties van organismen die gebonden zijn aan het water en die zich dus niet eenvoudig via lucht of land kunnen verspreiden, zoals meervallen (Vroom et al. 2016) of zoetwaterpissebedden (Elbrecht et al. 2018). Dergelijke soorten kunnen een vertekend beeld opleveren bij een DNA analyse. Zo werden bijvoorbeeld in het gebied van het Hoogheemraadschap van Rijnland al 14 MOTU’s aangetroffen die gelinkt konden worden aan de soort Asellus aquaticus, waarvan de sequenties onderling tot meer dan 10% van elkaar verschilden (Beentjes et al. 2019b). Wanneer wordt gewerkt met MOTU’s in plaats van met soorten, kan dat dus leiden tot een betere inschatting van de genetische biodiversiteit in een systeem. Dat kan een completer beeld geven van de ecologische waterkwaliteit, omdat genetische varianten van een soort (haplotypen) of soorten waarbinnen genetisch wel verschillen zijn, maar die morfologisch nog niet kunnen worden onderscheiden (cryptische soorten) verschillende responsen kunnen laten zien op bepaalde stressoren (Macher et al. 2016).
Soortidentificatie gebaseerd op DNA versus morfologie. Er zijn de afgelopen jaren verschillende onderzoeken gepubliceerd waarin de soortenrijkdom van onder andere vissen en macrofauna zoals bepaald met traditionele (morfologische) technieken en met DNA analyses met elkaar zijn vergeleken. Hoewel er vaak veel overlap wordt aangetoond tussen de twee resulterende lijsten, is het interessant om de verschillen te kunnen verklaren. Veel van de verschillen ontstaan doordat er een verschil is in de resolutie van de taxonomie tussen de twee methoden. Voor macrofauna zijn bepaalde groepen op basis van morfologie lastiger te onderscheiden dan met DNA, bijvoorbeeld een soortenrijke groep als de dansmuggen. Met een gedegen DNA referentiedatabank kunnen de larven veel eenvoudiger op soortniveau worden onderscheiden. Bij het gebruik van DNA speelt ook de variatie in niveaus van kennis bij het identificeren van soorten geen rol meer. Grootschalig onderzoek heeft uitgewezen dat identificaties van taxa in een macrofauna monster tot wel 30% kunnen verschillen wanneer hetzelfde monster door verschillende analisten wordt onderzocht (Haase et al. 2010). DNA analyses die gebruik maken van dezelfde referentiedatabank kunnen ook beter met elkaar worden vergeleken. Aan de andere kant zijn er ook groepen die morfologisch makkelijker te onderscheiden zijn dan met DNA, omdat er soms niet genoeg resolutie in de gebruikte DNA barcode sequenties zit om twee soorten goed uit elkaar te houden. Zo is de standaard DNA barcode voor het gewoon bootsmannetje (Notonecta glauca) identiek aan die van het zwart bootsmannetje (N. obliqua). Andere verschillen tussen morfologie en DNA analyse worden verklaard door het ontbreken van soortenmateriaal in de referentiedatabanken, of het achterlaten van te weinig eDNA in de omgeving.
Replica monsters. Om een representatief beeld te krijgen van de soortensamenstelling van een waterlichaam is het belangrijk om ervoor te zorgen dat er meerdere replicamonsters worden genomen op verschillende punten, zoals besproken in hoofdstuk 6. Dit hangt samen met de staat en het transport van het eDNA (zie hoofdstuk 5), welke er beide voor kunnen zorgen dat eDNA niet gelijkmatig is verdeeld in de waterkolom. Naast ruimtelijke variatie is de variatie in de tijd (bijvoorbeeld tussen seizoenen) ook een belangrijke bron van onderlinge verschillen tussen monsters, zeker waar het micro-organismen betreft (Beentjes et al. 2019a). Op grotere schaal geldt dit ook voor de verschillende samenstelling van eDNA monsters in verschillende seizoenen, zoals bij insecten die slechts een deel van hun levenscyclus in de waterkolom doorbrengen. Als locaties of tijdstippen niet zorgvuldig worden gekozen, dekken de soortenlijsten die voortvloeien uit NGS analyses niet de volledige soortensamenstelling. Dit alles geldt overigens niet alleen voor resultaten gebaseerd op een DNA bemonstering, maar ook voor traditionele monitoring. De lagere kosten van DNA analyses ten opzichte van traditionele monitoring bieden echter wel de mogelijkheid om meer monsters (zowel ruimtelijk als temporeel) te kunnen analyseren (zie “Kostenefficiëntie” in hoofdstuk 8).
Standaardisatie. DNA technieken worden binnen de onderzoekswereld wel breed toegepast, maar ze zijn niet zonder meer een op een inpasbaar in de huidige (op traditionele monitoring resultaten gebaseerde) maatlatten voor de ecologische kwaliteitsbeoordeling via de KRW. Wil men op den duur (delen van) de voor de KRW benodigde informatie vervangen of aanvullen met gegevens afkomstig uit DNA technieken, dan zal daar ook enige standaardisatie en intercalibratie nodig zijn. De resultaten van diverse methoden die gebruikt worden door verschillende instituten kunnen nogal van elkaar afwijken, door de keuzes die gemaakt worden in het traject van bemonstering tot analyse (zie hoofdstuk 6). Het lijkt, zeker op breder Europees niveau, lastig om alle stappen in het DNA proces volledig vast te leggen in een standaard protocol. Dat is ook met de huidige traditionele waterkwaliteitsmonitoring in Europa niet gebeurd en het is daar ook niet noodzakelijk gebleken.
KRW-monitoring is echter maar één van de vele manieren om naar watersystemen te kijken. DNA technieken bieden een bruikbaar alternatief voor allerhande biologische kwaliteitsbeoordelingen van watersystemen, waaronder veel analyses waar voor de huidige gebruikte methodieken ook geen standaarden zijn voorgeschreven. Denk hierbij aan het beoordelen van herstel- en onderhoudswerkzaamheden, maatregel-effect analyses, of het opsporen van exoten. Om resultaten met elkaar te kunnen vergelijken, zal zich echter wel een consensus moeten ontwikkelen over de rapportage van de stappen in het proces, de vergelijkbaarheid van resultaten en de controles die op de verschillende punten worden meegenomen. Onder meer om toe te werken naar een dergelijke consensus is in de afgelopen jaren een Europees platform opgericht onder de naam DNAqua-Net, waarin verschillende instituten die zich bezighouden met (e)DNA in samenwerking met beleidsmakers en andere stakeholders samen proberen toe te werken naar een “gouden standaard” voor DNA technieken (Leese et al. 2016).
Abundantie. Met methoden als qPCR of ddPCR kunnen zeer nauwkeurig eDNA concentraties in een monster worden bepaald, maar om die informatie vervolgens te vertalen naar de hoeveelheid biomassa, of naar het aantal individuen is niet eenvoudig en het vraagt de nodige soort-specifieke calibratie en validatie studies onder diverse omstandigheden. Er zijn verscheidene manieren waarop de concentratie van eDNA wordt beïnvloed door externe factoren. De snelheid van het afscheiden van eDNA hangt af van de soort en de tijd van het jaar, en de afbraak van eDNA (zie ook hoofdstuk5) hangt af van onder andere temperatuur, pH, bacteriële activiteit, hoeveelheid UV straling, en hoeveelheid organisch materiaal (waar DNA aan kan binden). De gemeten eDNA concentraties kunnen wél gebruikt worden om vergelijkingen in termen van DNA hoeveelheden te maken over de tijd of tussen locaties. Het is dan wel belangrijk om genoeg replica monsters te nemen om een betrouwbare schatting te doen van de werkelijke concentratie, rekening houdend met de ruimtelijke variatie die altijd aanwezig is in een waterlichaam. Niettemin blijft een directe koppeling van de gemeten DNA hoeveelheden met aantallen of biomassa niet gemakkelijk. Deze resultaten dienen vooral als relatief eenvoudig verkrijgbare informatie over mogelijke verschillen in abundanties tussen locaties en door de tijd.
Het verkrijgen van abundantie data is zeer lastig bij gebruik van metabarcoding, zeker met eDNA als input. Voor metabarcoding geldt dat op verschillende punten in het proces van bemonsteren en de verwerking in het lab de verhouding tussen DNA hoeveelheden van verschillende soorten kunstmatig kan worden beïnvloed. De voornaamste reden is de zogenaamde PCR bias, waar DNA van sommige soorten preferentieel wordt geamplificeerd ten opzichte van andere soorten, omdat de gekozen primers beter passen op de betreffende sequenties. Bij analyses waar wordt gekeken naar veranderingen door de tijd (maatregel-effect analyses) hoeft de PCR bias niet noodzakelijk een probleem te zijn (zie bijvoorbeeld Beermann et al., 2018). Het is echter raadzaam om de metabarcoding resultaten om te zetten naar “aangetoond/niet aangetoond”, in plaats van met de abundantie van de DNA fragmenten in een monster verder te werken. Een tweetal recente onderzoeken heeft overigens aangetoond dat abundantie van beperkte invloed is voor standaard ecologische kwaliteitsratio (EKR) beoordelingen van macrofauna (Beentjes et al. 2018, Buchner et al. 2019). Dat neemt niet weg dat kwantitatieve data alsnog belangrijk zijn voor andere analyses met betrekking tot de waterkwaliteitsbeoordeling, bijvoorbeeld bij vissen. Naast het feit dat het onmogelijk is om sequentie-aantallen uit een metabarcoding analyse te vertalen naar een hoeveelheid exemplaren, kan een eDNA analyse ook (nog) geen inzicht geven in de leeftijd, het formaat of het geslacht van vissen. Voor dergelijke kennisvragen zal eDNA niet een vervangende oplossing bieden. De vraag is of de voor waterbeheer benodigde informatie over de waterkwaliteit niet even goed kan worden verkregen met alternatieve maatlatten gebaseerd op DNA gegevens. Daarnaast kan eDNA ook ingezet worden in de vorm van een soort “quick scan”, waarbij de eDNA resultaten sturing kunnen geven aan het uitvoeren van uitgebreider onderzoek op gerichte plaatsen.
“Big data”. Een grote uitdaging voor het gebruik van NGS technieken is de hoeveelheid data die uit een analyse komt. In de toekomst wordt de hoeveelheid data alleen nog maar groter. Naast de computerkracht die nodig is om deze data op een redelijke termijn te laten analyseren, is ook veel opslagcapaciteit nodig om de data voor later gebruik op te slaan. Daarnaast is het echter ook zo dat meer data niet noodzakelijkerwijs betekent dat er betere resultaten uit een analyse komen. De vraagstelling die vooraf gaat aan een dergelijke analyse zal altijd leidend moeten zijn. Het is meestal beter om naar een aantal specifiek gedefinieerde delen van een gemeenschap te kijken en deze als indicatoren te gebruiken voor de ecologische toestand en het functioneren van het systeem, dan om te proberen om met DNA naar het totale palet aan biodiversiteit in een systeem te kijken. Enerzijds omdat hele brede DNA detectie methoden meestal veel bias vertonen in het voordeel van bepaalde taxonomische groepen, anderzijds omdat sommige groepen ecologisch meer relevante informatie opleveren dan andere. Er zijn wel methoden om uit een dataset met veel onbekende MOTU’s (die niet direct gelinkt kunnen worden aan een soort) informatie te verkrijgen die relevant kan zijn voor waterkwaliteits-beoordelingen, maar deze worden nog volop onderzocht in lopende studies (zie ook “Taxonomie-vrije methoden” in hoofdstuk 8).
8. Ontwikkelingen op het gebied van DNA
Kostenefficiëntie. Met het opschalen van DNA technieken zullen deze steeds goedkoper worden, hetgeen zeker bij gebruik van eDNA voor bepaalde taxonomische groepen een grote besparing kan opleveren ten opzichte van de huidige methodiek. Soortspecifieke detectie via watermonsters kan tot tien keer goedkoper zijn dan het gebruik van traditionele vangmethoden (Biggs et al. 2015, Sigsgaard et al. 2015). DNA methoden worden bovendien per monster goedkoper wanneer grotere hoeveelheden tegelijk geanalyseerd worden, omdat veel laboratorium werkzaamheden goed opgeschaald kunnen worden. Een pilotstudie kan soms nog een redelijke investering vragen, maar vaak hangen die kosten samen met het (verder) ontwikkelen van een methode, of omdat analyses meer tijd vergen om meerdere methoden met elkaar te vergelijken. Wanneer een methode (zoals een soort-specifieke PCR) eenmaal bestaat, hoeft een dergelijk validatie-traject niet meer te worden doorlopen.
Wanneer DNA technieken goedkoper worden dan traditionele monitoring, kan zich dat vertalen in de mogelijkheid om meer locaties te bemonsteren of om op meer tijdspunten te bemonsteren, om zo meer inzicht te krijgen in de variatie tussen verschillende seizoenen. Door vaker of op meer locaties te bemonsteren, is het mogelijk om een brede “quick scan” analyse te doen, en vervolgens op de plaatsen waar veranderingen gesignaleerd worden, met meer uitgebreide (wellicht deels traditionele) technieken aan de gang te gaan om een beter inzicht te krijgen in de probleemgebieden. Maatregel-effect analyses zouden eveneens kunnen profiteren van snellere en goedkopere eDNA technieken, omdat vaker en op meer locaties meten een betere resolutie geeft om veranderingen in een systeem te beoordelen.
Nieuwe bio-indicatoren. Alle organismen bevatten DNA, het is daarom mogelijk om middels DNA inzicht te krijgen in taxonomische groepen die normaliter buiten het bereik van de traditionele monitoring lagen, omdat deze bijvoorbeeld niet op basis van morfologische kenmerken te onderscheiden zijn. Denk aan bacteriën, schimmels en grote delen van het fyto- en zooplankton die lastig morfologisch te herkennen zijn. Daarnaast is gebleken dat voor groepen die al redelijk bekend zijn, zoals macrofauna, er nog sprake kan zijn van cryptische diversiteit (zie ook “Genetische variatie” in hoofdstuk 7). In de groepen waar de taxonomie en leefwijze nog grotendeels onbekend zijn, zullen deze inzichten lastig te vertalen zijn naar een kwaliteitsindicatie van een waterlichaam op basis van de kenmerken van de organismen. Mogelijk is het nodig om op den duur kwaliteitsindexen te herzien of nieuwe indexen te ontwikkelen die beter aansluiten bij de informatie die wordt verkregen vanuit DNA methoden (Pawlowski et al. 2018). De huidige maatlatten zijn ingericht om rekening te houden met de beperkingen van bestaande morfologische analyses, en daardoor lastig direct met resultaten gebaseerd op DNA technieken te vergelijken en in te vullen.
Taxonomie-vrije methoden. Omdat steeds meer onderzoek wordt gedaan met (e)DNA analyses, wordt er in rap tempo steeds meer informatie ingewonnen over genetische samenstelling van soorten en soortcomplexen. Grootschalig onderzoek, zoals het Global Malaise Trap Program (https://biodiversitygenomics.net/projects/gmp/), laat daarnaast zien dat er nog veel onbeschreven soorten zijn, waarvoor de klassieke taxonomische beschrijvingen op zich laten wachten. Om beter gebruik te kunnen maken van de enorme onbeschreven diversiteit, met name bij micro-organismen, wordt momenteel onderzoek gedaan naar methoden voor waterkwaliteitsbeoordeling die los staan van taxonomische namen en soortenlijsten. Deze methoden op basis van MOTU’s maken gebruik van “machine learning” technieken. Door van een grote hoeveelheid locaties met bekende kwaliteitseigenschappen zogenaamde DNA profielen te genereren, kan onderzocht worden of bepaalde MOTU’s indicatief zijn voor bepaalde eigenschappen van het water. Vervolgens kan een watermonster van een te onderzoeken locatie worden vergeleken met bestaande profielen, om zo, zonder tussenkomst van een soortenlijst, een kwaliteitsindicatie te doen voor het water. Pilot studies hebben al laten zien dat patronen van MOTU’s die aanwezig zijn in de waterkolom met redelijke nauwkeurigheid een indicatie kunnen geven van de status van verontreiniging (Li et al. 2018). Ook voor diatomeeën, een belangrijke indicatorgroep, is gebleken dat de zogenaamde “taxonomie-vrije” methoden betere beoordelingen opleverden (Cordier et al. 2018).
Levend versus dood. Een van de vragen die regelmatig opkomt bij onderzoeken met eDNA gaat over het vermogen om een onderscheid te maken tussen een actueel en een historisch signaal. Wanneer eDNA van een soort gevonden wordt, hoeft dat niet altijd te betekenen dat de soort op dat moment ook daadwerkelijk aanwezig was, wellicht is het nog eDNA van een week of een maand geleden. De staat van eDNA (zie hoofdstuk 5) zal er echter in de meeste gevallen voor zorgen dat, in ieder geval in de waterkolom, een eDNA signaal een redelijk accuraat beeld geeft van het moment van bemonsteren. Afhankelijk van de condities in het sediment (zuurstof, temperatuur, pH, bacteriële activiteit) kan DNA daarin tot maanden detecteerbaar blijven, ook na het verdwijnen van soorten (Turner et al. 2015). Hierdoor geldt dat voor resultaten uit sedimentmonsters niet altijd uitgegaan kan worden van een actueel signaal. Naast het verschil tussen actueel en historisch materiaal, kan er ook mogelijk vraag zijn naar onderscheid tussen levend en dood materiaal. Het eDNA kan immers ook afkomstig zijn van dode organismen. Dit kan een probleem zijn voor bijvoorbeeld genetisch analyse van invasieve exoten in havens. Als ballastwater wordt behandeld om organismen te doden voordat het geloosd wordt, komt er alsnog DNA van deze organismen in het water terecht. Een deel van dit probleem kan worden verholpen door regelmatig te monitoren. Invasieve exoten laten continue eDNA los in de omgeving, dood materiaal uit een lozing van ballastwater is een eenmalig signaal. Recent zijn ook onderzoeken gedaan op het gebied van “environmental RNA” (eRNA) (Cristescu 2019). RNA is een vorm van enkelstrengs-DNA dat alleen wordt gegeneerd door levende cellen. Omdat RNA enkelstrengs is, in tegenstelling tot het dubbelstrengs DNA, is het zeer instabiel en wordt het snel afgebroken in de cellen. Aanwezigheid van eRNA zou daarom gebruikt kunnen worden voor het aantonen van levende organismen. Dit veld van onderzoek is nog vrij nieuw, maar de eerste resultaten zijn voorzichtig positief (Pochon et al. 2017, Laroche et al. 2018)
9. Conclusies en aanbevelingen
DNA technieken kunnen breed ingezet worden voor de in het waterbeheer noodzakelijke informatievoorziening over biodiversiteit en omwikkelingen in ecologische waterkwaliteit. Er is de laatste jaren veel onderzoek verricht aan met name de metabarcoding en het gebruik van (e)DNA. Technieken zijn daardoor steeds robuuster en nauwkeuriger geworden, waardoor ze makkelijker zijn in te zetten bij reguliere monitoring. Het is daarbij wel belangrijk dat waterbeheerders op de hoogte zijn van de mogelijkheden en de beperkingen van het gebruik van DNA, en dat onderzoeksvragen en gebruikte methodieken op elkaar worden afgestemd. Niet alle methodieken zijn geschikt voor alle typen monitoring. Bij de meeste NGS methoden worden voornamelijk de algemene soorten uitgelicht. Gebruik van metabarcoding technieken kan er toe leiden dat zeldzame soorten verloren raken in de achtergrond, omdat er te veel signaal afkomstig is van algemene soorten. Voor het monitoren van (verwachte of gezochte) zeldzame soorten is het dan ook beter om een gerichte analyse te gebruiken, zoals de qPCR/ddPCR, in plaats van een NGS.
Het gebruik van eDNA opent de deuren voor het analyseren van taxonomische groepen die eerder buiten bereik lagen van de watermonitoring, omdat ze met traditionele methoden om technische of economische redenen nauwelijks te analyseren waren. De kostenefficiëntie van DNA methoden, zeker bij toepassing in grote hoeveelheden, zal er ook toe leiden dat er op meer locaties en/of vaker in de tijd bemonsterd en gemonitord kan worden. Het ligt voor de hand dat DNA methoden ingezet gaan worden voor een brede en snelle monitoring van een beheergebied (een zogenaamde “quick scan”), waar indien nodig ingezoomd kan worden op probleem gebieden met een meer uitgebreide monitoring, waarbij ook traditionele methoden nog steeds belangrijk zullen zijn.
De komende jaren zal het onderzoek met eDNA en metabarcoding zich steeds meer gaan richten op de toepassingen, nu veel methodieken dusdanig doorontwikkeld zijn dat operationalisering geen verre toekomstmuziek meer is. Het is niettemin nog steeds belangrijk dat ook het fundamenteel onderzoek wordt doorgezet, om meer grip te krijgen op de vertaling van eDNA signalen naar daadwerkelijke ecologische interpretaties. Daarnaast zijn de meer praktijkgerichte onderzoeken ook van belang, waarmee onderzoekers en gebruikers een beter beeld krijgen van de verschillen die ontstaan door keuzes die gemaakt worden in het proces van bemonstering, verwerking en analyse.
Er lopen momenteel al diverse kleine en grote projecten in Nederland waarin eDNA en metabarcoding technieken worden toegepast. Het screenen van watermonsters voor invasieve of zeldzame soorten is in veel gevallen al succesvol gebleken. De bemonstering van water is veel minder verstorend en arbeidsintensief, en voor veel soorten zijn inmiddels specifieke qPCR essays beschikbaar. Metabarcoding van eDNA wordt momenteel nog in meer beperkte vorm toegepast, voornamelijk in projecten waarin traditionele vismonitoring wordt vergeleken met eDNA. Daarnaast is metabarcoding momenteel al een essentiële techniek voor het monitoren van micro-organismen zoals bacteriën, en andere groepen zoals fytoplankton tonen veel potentie. In de komende jaren zal het nodig zijn om, zeker voor de metabarcoding technieken, hieraan meer uitgebreid onderzoek te doen parallel aan traditionele monitoring (indien beschikbaar). Ook zullen waterbeheerders vertrouwd moeten raken met het analyseren en interpreteren van waterkwaliteit met behulp van op DNA gebaseerde informatie. Alleen op die manier kan de techniek zich bewijzen en laten zien dat deze veel voordelen biedt ten opzichte van de huidige monitoring. Niet alleen door de daling in kosten en benodigde arbeid, maar ook door de enorme hoeveelheid informatie die kan worden gewonnen uit een watersysteem via environmental DNA.

 English resume
English resume